Microbioma Rizosférico de Bacterias en Maíz Criollo de Grano: Impacto Sobre el Rendimiento Bajo Transición Agroecológica
DOI:
https://doi.org/10.28940/terra.v41i0.1664Palabras clave:
Acinetobacter, Moroxellaceae, Proteobacteria, secuenciación, SphingobacteriumResumen
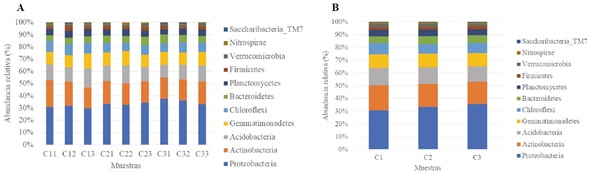
El maíz es un cereal de importancia en términos de nutrición humana y animal a nivel mundial. En el presente estudio se comparó el microbioma bacteriano de la rizosfera del suelo entre tres razas criollas de maíz (Zea mays L.), forrajero (C1), doble propósito (C2) y grano (C3); asimismo se determinó su impacto sobre el rendimiento. Se trabajó en parcelas ubicadas en el Campo Experimental El Retiro de la Universidad Autónoma Agraria Antonio Narro (UAAAN). El muestreo de suelo se efectuó el 13 de junio del 2021, 51 días después de la siembra. El DNA se extrajo a partir de tres muestras de cada tipo de raza de maíz. Se amplificó la región V3-V4 del gen 16S rRNA y se realizó secuenciación masiva de siguiente generación con Illumina; el análisis bioinformático se desarrolló en QIIME utilizando la referencia taxonómica bacteriana EzBioCloud. El phylum Proteobacteria, la clase Phycisphaerae, los órdenes Shingomonadales, Micrococcales y Phycisphaerales, las familias Moroxellaceae, Micrococcaceae, Chitinophagaceae, Shingobacteriaceae y Enterobacteriaceae, así como los géneros Acinetobacter, Shingobacterium y Cryseobacterium, fueron los taxa bacterianos que difirieron en abundancia relativa entre las tres razas (P < 0.05, prueba Kruskal-Wallis y una disimilitud promedio > 0.10 en el análisis SIMPER). Dentro de los taxa mencionados se detectaron bacterias promotoras de crecimiento vegetal (BPCV) como Acinetobacter y Sphingobacterium. Se concluye que, de las razas analizadas, la C2 mostró los mejores rendimientos de peso del grano (15.56 Mg ha-1) y peso seco en forraje (10.61 Mg ha-1), la cual representa una buena opción para preservar la semilla y realizar investigaciones posteriores bajo diferentes condiciones.
Descargas
Publication Facts
Reviewer profiles N/D
Author statements
- Academic society
- Terra Latinoamericana